RDkit
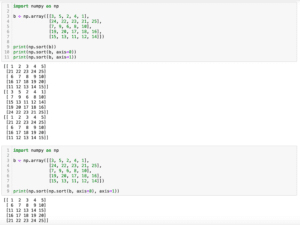
前回、PythonのNumPyでndarrayの要素をソートするsort関数を紹介しました。
今回は化合物を扱うためのライブラリです。
化合物を取り扱う研究者にとって面倒なのがパワーポイントなどで化合物の分子構造を見せること。
一つの化合物でもいくつか描き方があって、あっちこっちのサイトから画像を持ってくると意外と違う書き方がしてあってプレゼンテーションに統一感がなくなってしまいます。
かといってパワーポイントで線を一本ずつ書いて描画するのは大変だし、綺麗に書けなかったりします。
もちろんお金のある人とかがっつり専門の人はChemDrawのようなソフトを買うのでしょうが、たまーにしか化合物の構造を見せる必要がない人にとってはそこまでお金をかけられない。
そんな人にとって助けとなるのが今回紹介するRDkitです。
オープンソースツールなのでお金も掛かりませんし、専門のウェブサイトから構造の情報を引っ張ってきて、このライブラリで処理するだけで綺麗に描画できます。
ということで始めていきましょう。
インストール
ライブラリのインストールはいつも通りpipから行えます。
pip install rdkit分子構造の描画
化合物の分子構造を描画するにはまずはその化合物の構造の情報が必要です。
構造の記述の仕方はいくつかある様ですが、今回はSMILES(Simplified Molecular Input Lie Entry System)を紹介します。
RDkitを使って分子構造を描画するには、RDkitの「Chem」をインポートします。
そして記述方式がSMILESの場合は「Chem.MolFromSmiles(SMILES)」とします。
グルコースを描いてみるとこんな感じです。
from rdkit import Chem
glucose_smiles = "OC[C@@H](O1)[C@@H](O)[C@H](O)[C@@H](O)[C@@H](O)1"
glucose = Chem.MolFromSmiles(glucose_smiles)
glucose
実行結果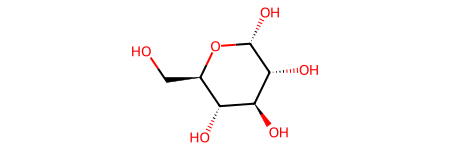
Jupyter notebookの場合、「Chem.MolFromSmiles(SMILES)」の返り値だけで分子構造が表示されます。
構造を画像として保存したい場合はさらに「Draw」をインポートして「Draw.MolToFile(構造, ファイルパス)」で保存できます。
from rdkit import Chem
from rdkit.Chem import Draw
glucose_smiles = "OC[C@@H](O1)[C@@H](O)[C@H](O)[C@@H](O)[C@@H](O)1"
glucose = Chem.MolFromSmiles(glucose_smiles)
Draw.MolToFile(glucose, "glucose.png")
実行結果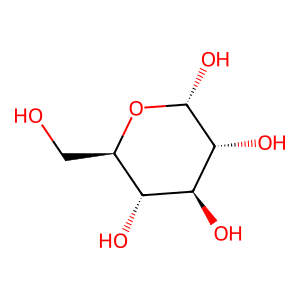
SMILESの記述方法
ここで問題はグルコースのSMILESでの記述がこの様な複雑なものであること。
"OC[C@@H](O1)[C@@H](O)[C@H](O)[C@@H](O)[C@@H](O)1"全くわからないでは困るので、ざっくりとSMILESがどういう記述方法なのか見ていきましょう。
単純に酸素「O」だけ描くと、水素「H」が付加されH2Oになります。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "O"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol1.png")
実行結果
水素「H」を明示的に描く時は「[H]」とします。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "[H]O[H]"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol2.png")
実行結果
二重結合は「=」で、また三重結合は「#」とします。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "O=C=O"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol3.png")
実行結果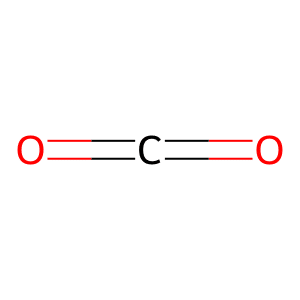
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "N#N"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol4.png")
実行結果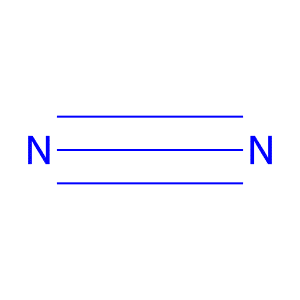
ナトリウム「Na」のように二文字で表される元素の場合は「[Na]」と記述します。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "[Na]"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol5.png")
実行結果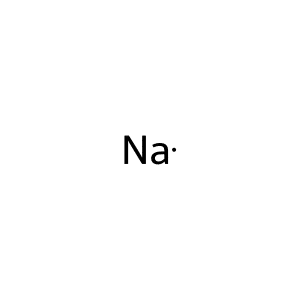
イオンにするには「+」や「-」を付けます。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "[Na+]"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol6.png")
実行結果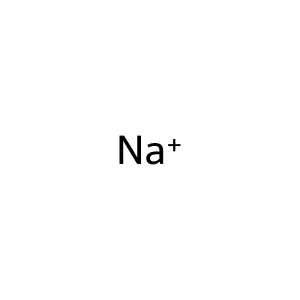
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "[Cl-]"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol7.png")
実行結果
続けて書けば二つの元素をつなぎ合わせることができます。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "[Na][Cl]"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol8.png")
実行結果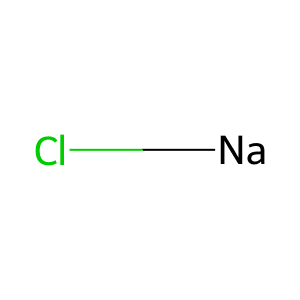
なぜか「[Na+][Cl-]」は表示できませんでした。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "[Na+][Cl-]"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol9.png")
実行結果
[22:22:37] Explicit valence for atom # 1 Cl, 2, is greater than permitted
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
Cell In[13], line 8
4 mol_smiles = "[Na+][Cl-]"
6 mol = Chem.MolFromSmiles(mol_smiles)
----> 8 Draw.MolToFile(mol, "mol9.png")
File /Library/Frameworks/Python.framework/Versions/3.10/lib/python3.10/site-packages/rdkit/Chem/Draw/__init__.py:287, in MolToFile(mol, filename, size, kekulize, wedgeBonds, imageType, fitImage, options, **kwargs)
285 raise ValueError('no filename provided')
286 if not mol:
--> 287 raise ValueError('Null molecule provided')
289 if imageType is None:
290 imageType = os.path.splitext(filename)[1][1:]
ValueError: Null molecule providedしかし「[Na+][OH-]」は表示できました。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "[Na+][OH-]"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol10.png")
実行結果
[22:23:19] unsupported number of radical electrons 7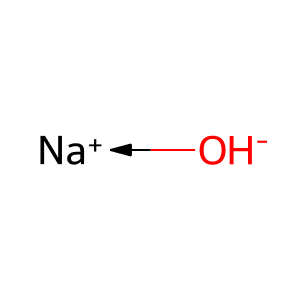
炭素「C」をつなげて描くと炭素鎖ができます。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "CCCCCC"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol11.png")
実行結果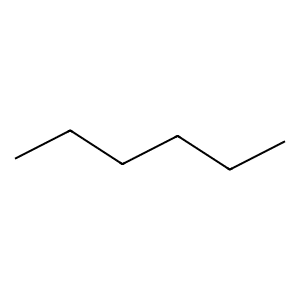
炭素2つに同じ番号をつけると繋げることができます。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "C1CCCCC1"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol12.png")
実行結果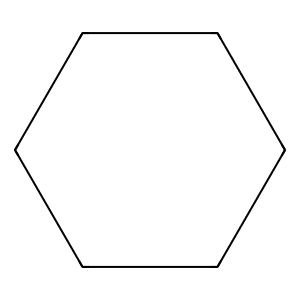
ベンゼン環(共鳴構造?)の場合は小文字の「c」にします。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "c1ccccc1"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol13.png")
実行結果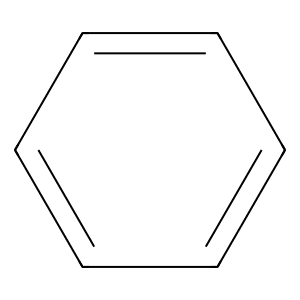
側鎖構造を見ていきましょう。
アラニンをSMILESで記述してみるとこうなります。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "N[CH](C)C(=O)O"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol14.png")
実行結果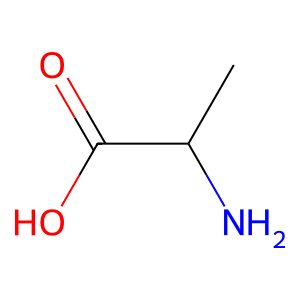
アミノ酸は光学異性体と言って、同じ原子で構成されていても結合の向きが変わることで違う性質を持つことがあります。
それを表示するには「@」や「@@」を用います。
「@」は構造の平面の向かって手前側に、「@@」は構造の平面の向かって奥側にその原子が位置します。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "N[C@H](C)C(=O)O"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol15.png")
実行結果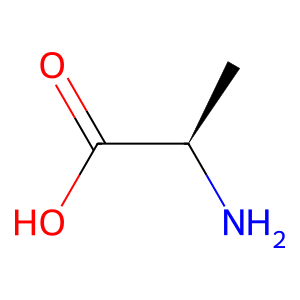
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "N[C@@H](C)C(=O)O"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol16.png")
実行結果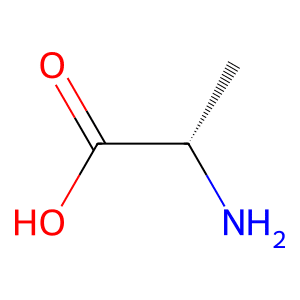
ここまで自分で構造情報を作って、構造を描画させる方法を紹介してきましたが、私の様にたまにしか構造式を必要としない人にとってはなかなか難しいと思います。
ということで次回はデータベースから構造情報を持ってきて、それを描画させる方法を紹介します。
ではでは今回はこんな感じで。

コメント