RDkit
前回、PythonのRDkitで化合物の分子構造を描画する方法を紹介しました。

今回はデータベースから化合物の構造情報を取得し描画する方法を紹介します。
前回学んだ通り、自分で化合物の構造情報を作り、描画するのは普段から使っていないとだいぶ難しいわけです。
そこでデータベースから化合物の構造情報を取得すれば、間違いもなく、かつ簡単に化合物の構造を描画できるというわけです。
今回は2つのデータベースから構造情報を取得する方法をグルコースを例に紹介します。
それでは始めていきましょう。
PubChem
まずはPubChemというデータベースです。

アクセスするとこんな画面になります。
真ん中の検索窓に「Glucose」と入れてエンターを押します。
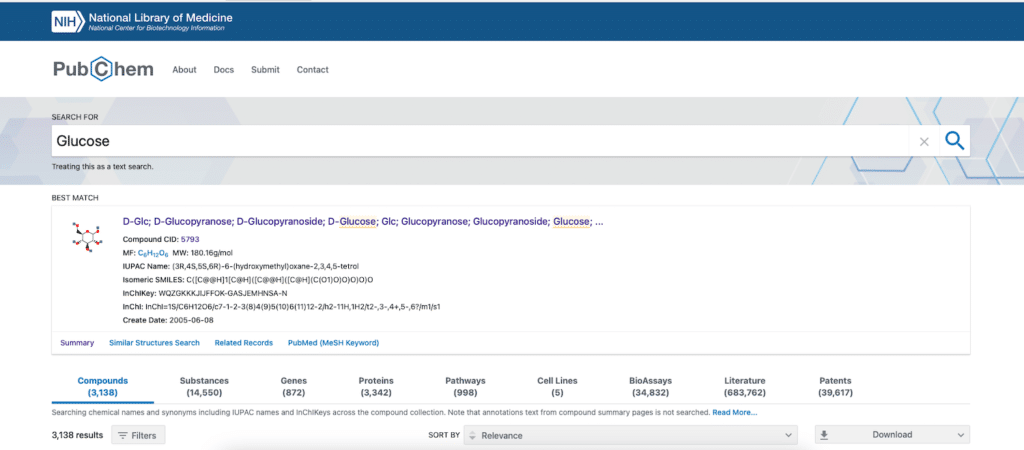
検索結果が表示されるので、構造を描画したい化合物を選択します。
今回はグルコース(Glucose)を検索して、一番マッチしたのが「D-Glc; D-Glucopyranose; D-Glucopyranoside; D-Glucose; Glc; Glucopyranose; Glucopyranoside; Glucose;…」となっていますが、これは別名が羅列されているだけで、どれもグルコース(光学異性体は注意が必要)ですので、化合物名をクリックします。
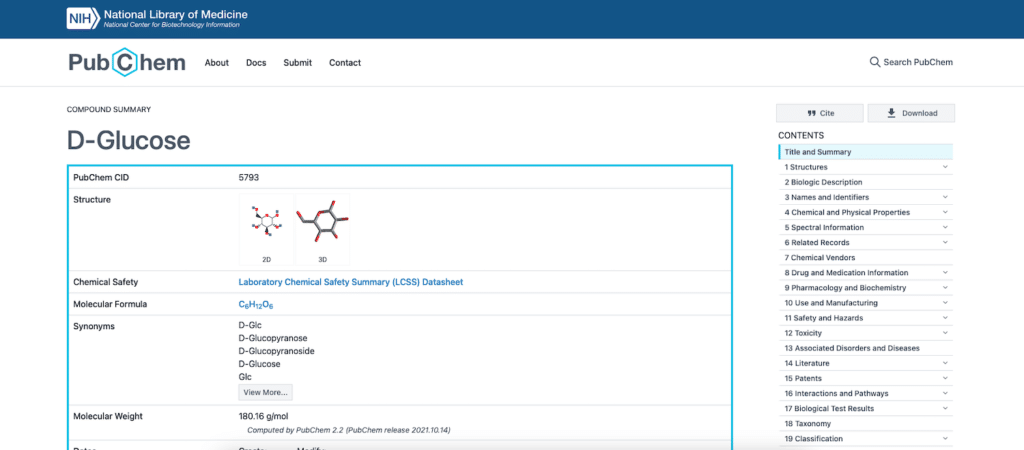
その物質のデータのページに移動します。
化合物の構造情報が記載されているのは「3 Names and Identifiers」というところなので、そこまで下に移動します。
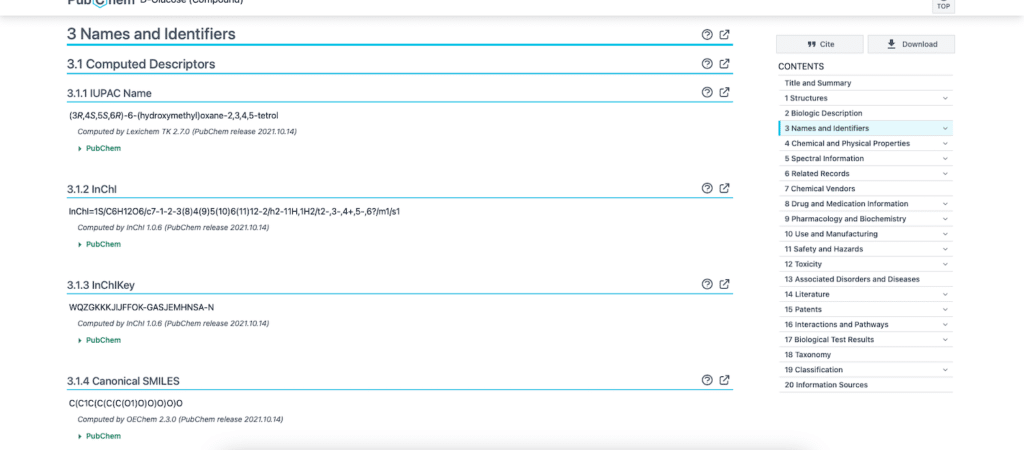
複数の記述の方法でグルコースの構造情報が記載されています。
今回はその中で「InChI」と「Canonical SMILES」、「Isomeric SMILES」を使って、構造を描画してみましょう。
SMILES
SMILESの構造情報から構造を描画する方法は前回お話ししましたが、「Chem.MolFromSmiles(smiles)」を用います。
今回、「Canonical SMILES」、「Isomeric SMILES」の2つの記述方式から構造を描画してみますが、この2つはこんな感じで違います。
#Canonical SMILES
C(C1C(C(C(C(O1)O)O)O)O)O#Isomeric SMILES
C([C@@H]1[C@H]([C@@H]([C@H](C(O1)O)O)O)O)Oただどちらの方式でもSMILESには変わりはないので、プログラムは同じです。
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "C(C1C(C(C(C(O1)O)O)O)O)O"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol1.png")
実行結果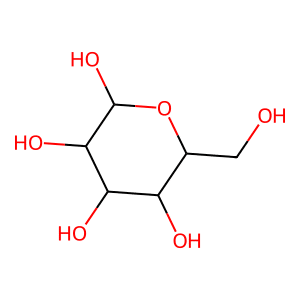
from rdkit import Chem
from rdkit.Chem import Draw
mol_smiles = "C([C@@H]1[C@H]([C@@H]([C@H](C(O1)O)O)O)O)O"
mol = Chem.MolFromSmiles(mol_smiles)
Draw.MolToFile(mol, "mol2.png")
実行結果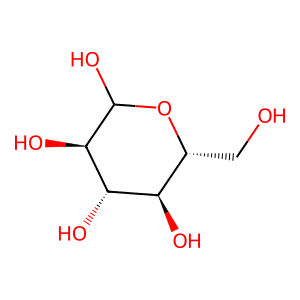
この2つぱっと見は同じ構造に見えますが、「Canonical SMILES」は分子は完全に平面で描かれているのに対し、「Isomeric SMILES」では分子の奥行き方向の情報まで含んで描かれています。
どちらが必要かはその状況で違うと思いますので、うまく使い分けてください。
InChI
次にInChIの記述方式の構造情報から化合物の構造を描画してみましょう。
PubChemのグルコースのページからInChIの記述方式の構造を取得します。
InChI=1S/C6H12O6/c7-1-2-3(8)4(9)5(10)6(11)12-2/h2-11H,1H2/t2-,3-,4+,5-,6?/m1/s1InChIの場合、構造を描画するには「Chem.MolFromInchi(InChI)」を用います。
from rdkit import Chem
from rdkit.Chem import Draw
mol_inchi = "InChI=1S/C6H12O6/c7-1-2-3(8)4(9)5(10)6(11)12-2/h2-11H,1H2/t2-,3-,4+,5-,6?/m1/s1"
mol = Chem.MolFromInchi(mol_inchi)
Draw.MolToFile(mol, "mol3.png")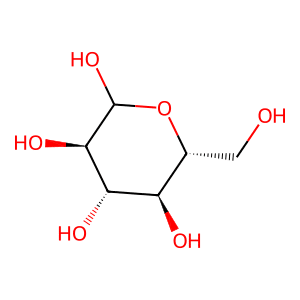
出力された構造は「Isomeric SMILES」と同じく、分子の奥行き方向の情報まで含んで描かれています
MERCK(Sigma-Aldrich)
化合物を購入する時、購入先の一つとして必ず名前が上がるだろうMERCK(メルク)社。
私の世代の人間には「Sigma-Aldrich(シグマアルドリッチ)」と言った方がピンとくるというと世代がバレてしまうのではないでしょうか。
MERCK社のウェブサイトから化合物を検索した際にも構造情報を取得できるので、その方法を紹介します。
ちなみに先ほどのPubChemでは英語でしか検索できませんが、MERCK社のウェブサイトでは日本語でも検索できるため、こちらの方がとっつきやすいかもしれません。
まずはメルク社のウェブサイトにアクセスし、上の検索窓に化合物名を入力します。
今回もグルコースを例に見ていきましょう。
購入したい商品が見つかったら、製品番号をクリックします。
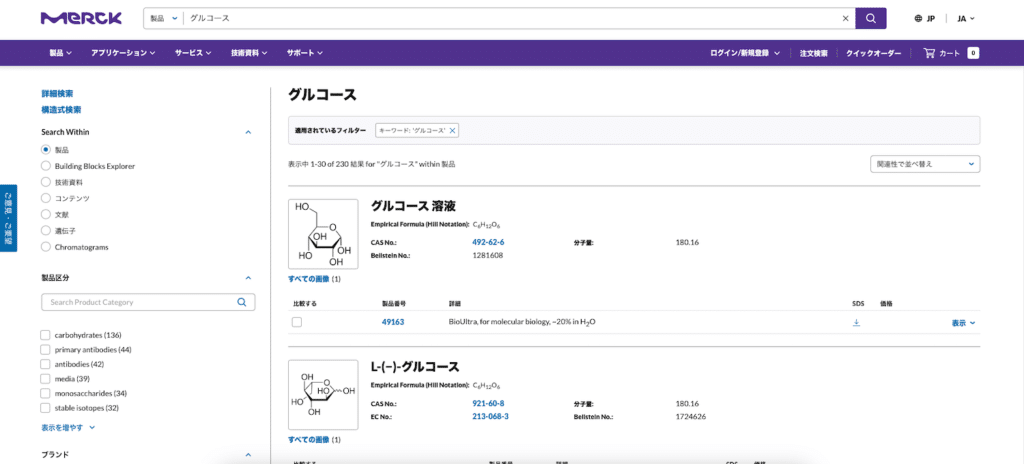
その製品の詳細ページに移動しますので、下にスクロースして「特性」の項目の一番最後までいきます。
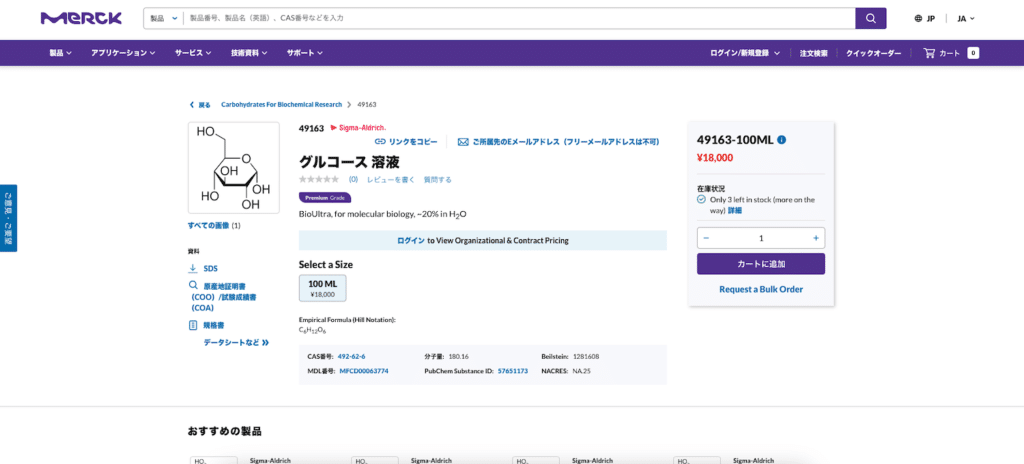
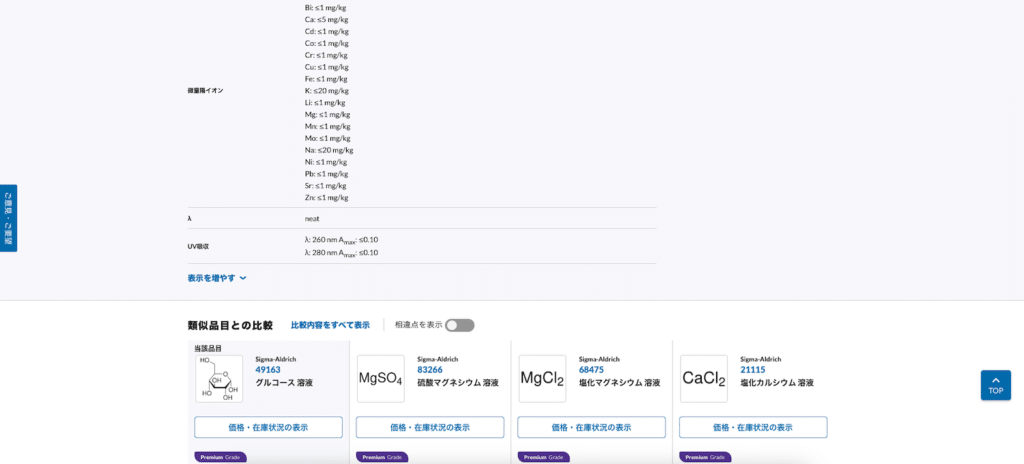
一番最後に「表示を増やす」があるのでクリックします。
クリックすると「SMILES string」と「InChI」があります。
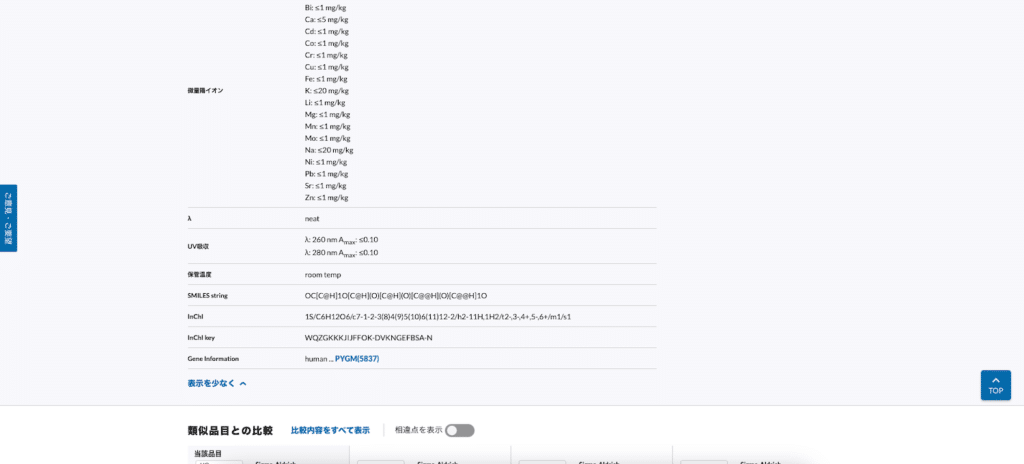
こんな感じで構造情報を取得して、RDkitで描画することで化合物構造を見せるのも苦じゃなくなりそうです。
次回はSciPyのintegrate.quadを使って積分する方法を紹介します。
ではでは今回はこんな感じで。

コメント