Apple Watch
前回、Apple Watchで心電図データを取得し、CSVファイルとして書き出しました。
今回は取得した心電図のデータをPythonのMatplotlibを使ってグラフとして表示してみましょう。
ちなみに今回は例としてこちらのデータを使用します。
誕生日等のデータを伏せてありますので、ご自由にお使いください。
それでは始めていきましょう。
ライブラリのインポートとファイルパスの設定
まずはライブラリのインポートとファイルパスの設定を行います。
ということでこんな感じ。
import os
import csv
ecg_filename = 'ecg_example.csv'
default_dirpath = os.getcwd()
ecg_filepath = os.path.join(default_dirpath, ecg_filename)
print(default_dirpath)
print(ecg_filepath)
実行結果
/Pythonプログラムのあるフォルダまでのパス
/Pythonプログラムのあるフォルダまでのパス/ecg_example.csvインポートするライブラリは「os」と「csv」です。
この書き方は最近の私の中の流行りで、基準点となるフォルダのパスを決め、そこから出入力のフォルダ名やファイル名をつなげて、フォルダやファイルまでのパスを作るという方法を使っています。
この点で良いのは、ファイル名やフォルダ名、または場所を変えた時に、どの部分を変えれば良いのか分かりやすいという点です。
といってもここら辺は好みでいいのかなと思います。
CSVファイルの読み込み
次に心電図のデータが入ったCSVファイルを読み込みます。
skiprows = 13
def ecgdataget():
data = []; samplerate = 512
with open(ecg_filepath, 'r', encoding='utf-8') as f_in:
ecg_file = csv.reader(f_in)
for i, row in enumerate(ecg_file):
if 'サンプルレート' in row:
samplerate = int(row[1].rstrip('ヘルツ'))
if i >= skiprows:
data.append(float(row[0]))
return data, samplerate
data, samplerate = ecgdataget()
print(samplerate)
print(data)
実行結果
512
[33.465, -11.283, -50.495, -81.613, -103.542, -117.064, -123.013, -122.754, -118.575,
-112.175, -105.136, -99.503, -96.304, -95.57, -97.256, -100.642, -104.577, -108.615,
-112.41, -115.382, -117.412, -118.547, -118.832, -118.735, -118.831, -119.314, -120.243,
-121.61, -123.237, -125.009, -126.835, -128.484, -129.682, -130.214, -129.931, -128.784,
-126.799, -123.984, -120.324, -115.825, -110.527, -104.524, -97.951, -90.957, -83.681,
(以下略)ここで重要なのはどのデータを取得し、どのデータを取得しないか決めることです。
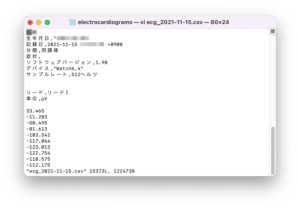
心電図データの入っているCSVの最初にはこんな感じで心電図データ以外の情報が含まれています。
名前
生年月日,"XXXX/XX/XX"
記録日,XXXX-XX-XX XX:XX:XX +0900
分類,洞調律
症状,
ソフトウェアバージョン,1.90
デバイス,"Watch6,4"
サンプルレート,512ヘルツ
リード,リードI
単位,µV実際、これからデータを解析・表示していく上でほとんどの情報は必要ありません。
ログを残すという意味では「記録日」、「分類」(解析結果)はあってもいいかもしれませんが、グラフの表示には必要ありません。
データの解析・表示だけに限って言えば「サンプルレート」は必要ですが、これは多分固定なので、この情報から取得してもしなくてもいいでしょう。
ただ今回は練習のためにも、サンプルレートはとりあえずこのCSVファイルから取得してみようと思います。
ということで上から解説していきましょう。
最初に「skiprows = 13」とありますが、これはファイルの先頭行から読み飛ばす行数のことです。
先ほどの心電図データの前の情報の部分が13行あるので、ここを読み飛ばすための数というわけです。
次にファイルを読み込みます。
def ecgdataget():
data = []; samplerate = 512
with open(ecg_filepath, 'r', encoding='utf-8') as f_in:
ecg_file = csv.reader(f_in)ここではまず心電図データを入れるための空のリストを作成し、サンプルレートを定義しています。
もしかしたらサンプルレートが何らかの理由で記載されていなかったり、読めなかったりした場合はデフォルトとして「512」ヘルツであると定義しています。
そして「with open(ecg_filepath, ‘r’, encoding=’utf-8′) as f_in:」で文字コードをutf-8としてファイルを読み込み、「ecg_file = csv.reader(f_in)」でCSV形式として読み込んでいます。
次に1行ずつ読み込み、if文を使って条件に合う行を抽出、データを取得していきます。
for i, row in enumerate(ecg_file):
if 'サンプルレート' in row:
samplerate = int(row[1].rstrip('ヘルツ'))
if i >= skiprows:
data.append(float(row[0]))
return data, samplerateまず「for i, row in enumerate(ecg_file):」で行番号と1行ずつのデータを取得していきます。
ちなみにenumerate関数に関しては、こちらで紹介していますので、よかったらどうぞ。

そして行に「サンプルレート」という言葉があった場合(if ‘サンプルレート’ in row:)、その行のコンマで区切られた2つ目の文字列(row[1])を取得し、文字列の最後にあるヘルツを取り除き(.rstrip(‘ヘルツ’))、int(整数)に変換して、変数samplerateを上書きします。
またそれとは別に行数がskiprowsで定義された数以上になった時(if i >= skiprows:)には、コンマで区切られた1番目の文字列を取得し、float(小数)に変換したのち、リストdataに追加します(data.append(float(row[0])))。
これで心電図データをサンプルレートを取得したので、「return data, samplerate」でリストと値を返しています。
そして作成した関数を実行して、確認のため表示しています。
data, samplerate = ecgdataget()
print(samplerate)
print(data)時間のリストを作成
ここまではいいのですが、これからグラフ表示するのに欠けているデータがあります。
それはX軸で用いる「時間」のデータです。
ということでリスト内包表記を使って時間のリストを作成してみましょう。
time = [n/samplerate for n in range(0, len(data))]
print(time)
実行結果
[0.0, 0.001953125, 0.00390625, 0.005859375, 0.0078125, 0.009765625, 0.01171875, 0.013671875,
0.015625, 0.017578125, 0.01953125, 0.021484375, 0.0234375, 0.025390625, 0.02734375, 0.029296875,
(中略)
29.97265625, 29.974609375, 29.9765625, 29.978515625, 29.98046875, 29.982421875, 29.984375,
29.986328125, 29.98828125, 29.990234375, 29.9921875, 29.994140625, 29.99609375, 29.998046875]ちなみにリスト内包表記に関しては、こちらで紹介していますので、よかったらどうぞ。

実はここでサンプルレートが出てきます。
今回のデータは512ヘルツで取得したデータということで、1秒回に512回心臓からの電位のデータを取得しています。
そこで「range(0, len(data))」でデータの個数のリストを作り、for文を使って一つずつサンプルレートで割ってやれば(n/samplerate)、時間に変換することができます。
Apple Watchでの測定時間は30秒ですので、最初が0秒、最後が30秒(正確には29.998046875秒)となっているので、ちゃんと時間が取得できていることがわかります。
Matplotlibを使ってグラフ表示
次にMaplotlibを使ってグラフ表示をしていきます。
import matplotlib.pyplot as plt
fig = plt.figure()
plt.clf()
plt.plot(time, data)
plt.xlabel('Sec')
plt.ylabel(u'\u03bcV')
plt.tight_layout()
実行結果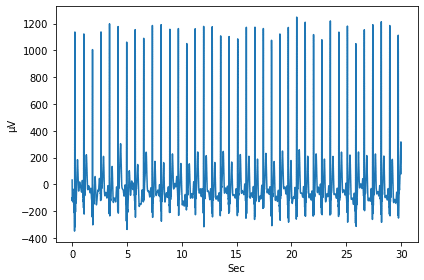
matplotlibに関しては色々と解説していますので、良かったら3PySci内を色々探してみてください。
ちなみに3PySci内でmatplotlibを最初に紹介している記事はこちらです。

今回は本当に単純なグラフにしています。
まずはグラフのプロットエリアを定義(fig = plt.figure())し、何度もグラフ表示した際、前のグラフが残っているのを防ぐため、一度クリア(plt.clf())しています。
そして「plt.plot(time, data)」でX軸を時間、Y軸を心電図データとしてプロットします。
X軸名として「Sec」(plt.xlabel(‘Sec’))、Y軸名として「μV」(plt.ylabel(u’\u03bcV’))を記載しています。
ちなみにY軸名の「u’\u03bcV’」のうち、「u’\u03bc’」はマイクロのユニコードでの表記です。
ここに関してはまた機会を設けて、紹介したいと思います。
今回はとりあえずそんなもんかと思ってもらえれば大丈夫です。
最後にグラフが描写エリアからはみ出ないように調整(plt.tight_layout())して完了です。
拡大してみる
先ほどの図ではどんな心電図になっているのか、もっと言うと健康診断で見かける心電図になっているのか分かりません。
ということで拡大してみましょう。
import matplotlib.pyplot as plt
fig = plt.figure()
plt.clf()
plt.plot(time, data)
plt.xlabel('Sec')
plt.ylabel(u'\u03bcV')
plt.xlim(0,2.5)
plt.tight_layout()
実行結果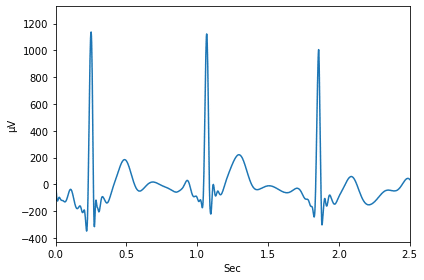
変わった部分は「plt.xlim(0,2.5)」が追加されたところです。
この「plt.xlim(最小値、最大値)」がX軸方向で範囲を指定するコマンドになります。
ちなみにY軸の場合は「plt.ylim(最小値,最大値)」です。
これでグラフ表示をすることができました。
次回はこのデータを使って、心臓の周波数成分の解析をフーリエ変換を使ってやってみたいと思います。
ではでは今回はこんな感じで。

コメント