Biopython
前回、Biopythonで相補鎖、反転配列、逆相補鎖、転写前後の配列、翻訳後の配列の取得方法を紹介しました。
今回はコドン表の表示と各生物のコドン表を使った翻訳方法を紹介します。
それでは始めていきましょう。
コドン表の表示
コドン表を表示するには「Bio.Data」の「CodonTable」をインポートします。
そして「CodonTable.表示するコドン表」とします。
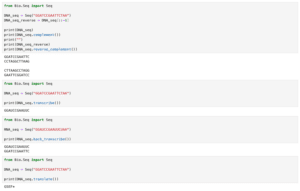
ここでは一般的に使われるコドン表「standard_dna_table」を表示してみます。
from Bio.Data import CodonTable
print(CodonTable.standard_dna_table)
実行結果
Table 1 Standard, SGC0
| T | C | A | G |
--+---------+---------+---------+---------+--
T | TTT F | TCT S | TAT Y | TGT C | T
T | TTC F | TCC S | TAC Y | TGC C | C
T | TTA L | TCA S | TAA Stop| TGA Stop| A
T | TTG L(s)| TCG S | TAG Stop| TGG W | G
--+---------+---------+---------+---------+--
C | CTT L | CCT P | CAT H | CGT R | T
C | CTC L | CCC P | CAC H | CGC R | C
C | CTA L | CCA P | CAA Q | CGA R | A
C | CTG L(s)| CCG P | CAG Q | CGG R | G
--+---------+---------+---------+---------+--
A | ATT I | ACT T | AAT N | AGT S | T
A | ATC I | ACC T | AAC N | AGC S | C
A | ATA I | ACA T | AAA K | AGA R | A
A | ATG M(s)| ACG T | AAG K | AGG R | G
--+---------+---------+---------+---------+--
G | GTT V | GCT A | GAT D | GGT G | T
G | GTC V | GCC A | GAC D | GGC G | C
G | GTA V | GCA A | GAA E | GGA G | A
G | GTG V | GCG A | GAG E | GGG G | G
--+---------+---------+---------+---------+--他にどのようなコドン表が収録されているかを見るには「CodonTable.unambiguous_dna_by_name」を用います。
from Bio.Data import CodonTable
tablename = CodonTable.unambiguous_dna_by_name
for name in tablename.keys():
print(name)
実行結果
Standard
SGC0
Vertebrate Mitochondrial
SGC1
Yeast Mitochondrial
SGC2
Mold Mitochondrial
Protozoan Mitochondrial
Coelenterate Mitochondrial
Mycoplasma
Spiroplasma
SGC3
Invertebrate Mitochondrial
SGC4
Ciliate Nuclear
Dasycladacean Nuclear
Hexamita Nuclear
SGC5
Echinoderm Mitochondrial
Flatworm Mitochondrial
SGC8
Euplotid Nuclear
SGC9
Bacterial
Archaeal
Plant Plastid
Alternative Yeast Nuclear
Ascidian Mitochondrial
Alternative Flatworm Mitochondrial
Blepharisma Macronuclear
Chlorophycean Mitochondrial
Trematode Mitochondrial
Scenedesmus obliquus Mitochondrial
Thraustochytrium Mitochondrial
Pterobranchia Mitochondrial
Candidate Division SR1
Gracilibacteria
Pachysolen tannophilus Nuclear
Karyorelict Nuclear
Condylostoma Nuclear
Mesodinium Nuclear
Peritrich Nuclear
Blastocrithidia Nuclear
Balanophoraceae Plastid
Cephalodiscidae Mitochondrialこの中から特定のコドン表を表示したい場合は「CodonTable.unambiguous_dna_by_name[“名前”]」とします。
from Bio.Data import CodonTable
print(CodonTable.unambiguous_dna_by_name["Standard"])
print(CodonTable.unambiguous_dna_by_name["Invertebrate Mitochondrial"])
実行結果
Table 1 Standard, SGC0
| T | C | A | G |
--+---------+---------+---------+---------+--
T | TTT F | TCT S | TAT Y | TGT C | T
T | TTC F | TCC S | TAC Y | TGC C | C
T | TTA L | TCA S | TAA Stop| TGA Stop| A
T | TTG L(s)| TCG S | TAG Stop| TGG W | G
--+---------+---------+---------+---------+--
C | CTT L | CCT P | CAT H | CGT R | T
C | CTC L | CCC P | CAC H | CGC R | C
C | CTA L | CCA P | CAA Q | CGA R | A
C | CTG L(s)| CCG P | CAG Q | CGG R | G
--+---------+---------+---------+---------+--
A | ATT I | ACT T | AAT N | AGT S | T
A | ATC I | ACC T | AAC N | AGC S | C
A | ATA I | ACA T | AAA K | AGA R | A
A | ATG M(s)| ACG T | AAG K | AGG R | G
--+---------+---------+---------+---------+--
G | GTT V | GCT A | GAT D | GGT G | T
G | GTC V | GCC A | GAC D | GGC G | C
G | GTA V | GCA A | GAA E | GGA G | A
G | GTG V | GCG A | GAG E | GGG G | G
--+---------+---------+---------+---------+--
Table 5 Invertebrate Mitochondrial, SGC4
| T | C | A | G |
--+---------+---------+---------+---------+--
T | TTT F | TCT S | TAT Y | TGT C | T
T | TTC F | TCC S | TAC Y | TGC C | C
T | TTA L | TCA S | TAA Stop| TGA W | A
T | TTG L(s)| TCG S | TAG Stop| TGG W | G
--+---------+---------+---------+---------+--
C | CTT L | CCT P | CAT H | CGT R | T
C | CTC L | CCC P | CAC H | CGC R | C
C | CTA L | CCA P | CAA Q | CGA R | A
C | CTG L | CCG P | CAG Q | CGG R | G
--+---------+---------+---------+---------+--
A | ATT I(s)| ACT T | AAT N | AGT S | T
A | ATC I(s)| ACC T | AAC N | AGC S | C
A | ATA M(s)| ACA T | AAA K | AGA S | A
A | ATG M(s)| ACG T | AAG K | AGG S | G
--+---------+---------+---------+---------+--
G | GTT V | GCT A | GAT D | GGT G | T
G | GTC V | GCC A | GAC D | GGC G | C
G | GTA V | GCA A | GAA E | GGA G | A
G | GTG V(s)| GCG A | GAG E | GGG G | G
--+---------+---------+---------+---------+--特定の生物のコドン表を使って翻訳する方法
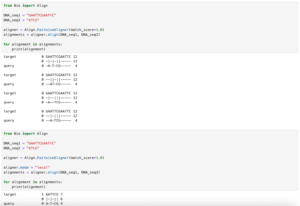
特定の生物のコドン表で翻訳するには、前回学んだ「配列.translate(table=”コドン表の名前”)」とします。
from Bio import SeqIO
for record in SeqIO.parse("AAA27722.1.fasta", "fasta"):
DNA_seq =record.seq
print(DNA_seq.translate())
print(DNA_seq.translate(table="Invertebrate Mitochondrial"))
実行結果
MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYG
VQCFSRYPDHMKQHDFFKSAMPEGYVQERTIFYKDDGNYKSRAEVKFEGDTLVNRIELKGIDFKEDG
NILGHKMEYNYNSHNVYIMADKQKNGIKVNFKIRHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYL
STQSALSKDPNEKRDHMILLEFVTAAGITHGMDELYK*
MSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTFSYG
VQCFSSYPDHMKQHDFFKSAMPEGYVQESTMFYKDDGNYKSRAEVKFEGDTLVNSIELKGIDFKEDG
NILGHKMEYNYNSHNVYIMADKQKNGIKVNFKISHNIEDGSVQLADHYQQNTPIGDGPVLLPDNHYL
STQSALSKDPNEKSDHMILLEFVTAAGITHGMDELYK*一般的なコドン表と「Invertebrate Mitochondrial」のコドン表を比べると「ATA」がイソロイシン(I)からメチオニン(M)に、「TGA」がストップコドン(Stop)からトリプトファン(W)に変わっています。
アライメントをしてみたら分かりやすいのですが、それは次回解説します。
今回はとりあえずメチオニン(M)とトリプトファン(W)の数を数えて違いがあるかみてみましょう。
特定のヌクレオチドやアミノ酸の数を数えるには「配列.count(“ヌクレオチドやアミノ酸”)」とします。
from Bio import SeqIO
for record in SeqIO.parse("AAA27722.1.fasta", "fasta"):
DNA_seq =record.seq
seq1 = DNA_seq.translate()
seq2 = DNA_seq.translate(table="Invertebrate Mitochondrial")
print(seq1.count("M"))
print(seq2.count("M"))
print(seq1.count("W"))
print(seq2.count("W"))
実行結果
7
8
1
1同じ配列を異なるコドン表で変換したところ、メチオニン(M)の数が一つ違っています。
次回はどこのアミノ酸が変わったのか見るために、ペアワイズアライメントの方法を紹介します。
ではでは今回はこんな感じで。

コメント