機械学習ライブラリScikit-learn
前回、機械学習ライブラリScikit-learnの糖尿病患者のデータセットを使い、SVRモデルのうちどのkernelでも共通に影響のある(だろう)オプションを試してみました。
今回はデフォルトのkernelであるrbfに影響があるオプションを試してみたいと思います。
ということで今回もデータの読み込みから。
<セル1>
from sklearn.datasets import load_diabetes
import pandas as pd
diabetes = load_diabetes()
df = pd.DataFrame(diabetes.data, columns=diabetes.feature_names)
df["target"] = diabetes.target
df
実行結果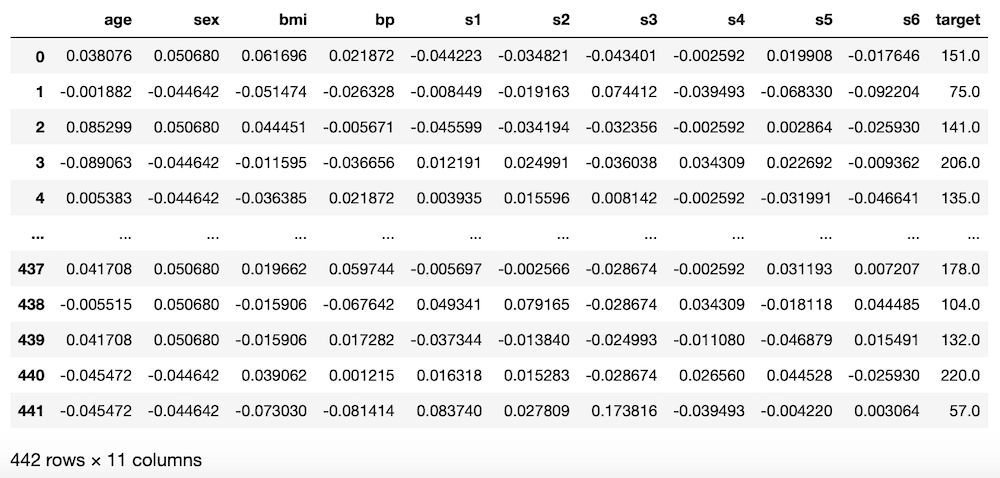
次に機械学習に用いる特徴量とターゲットをそれぞれ変数xとyに格納します。
(後で気付いたのですが、前の記事では「s6」も関連性がありそうということでしたが、忘れてしまいました。他の記事でも上記の組み合わせで行っているものもありますが、忘れたんだなぁと思って読んでください。)
<セル2>
x = df.loc[:, ["bmi", "s5", "bp", "s4", "s3"]]
y = df.loc[:, "target"]
実行結果ではSVRモデルを読み込んで、機械学習と予想を100回試した後、その予想精度のスコアの平均値を表示します。
今回はkernelをrbfにしますが、rbfはデフォルトなので、特に指定する必要はありません。
ただ後で見て分かりやすくするために、明示することにしましょう。
その際、過学習になっていないか確認するため、テスト用データでのスコアだけでなく、学習用データでのスコアも計算します。
<セル3>
from sklearn.model_selection import train_test_split
from sklearn.svm import SVR
from sklearn.metrics import r2_score
import numpy as np
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train_ori, x_test_ori, y_train_ori, y_test_ori = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR()
model_svr.fit(x_train_ori, y_train_ori)
pred_svr_ori = model_svr.predict(x_test_ori)
pred_svr_score.append(r2_score(y_test_ori, pred_svr_ori))
pred_svr_ori_train = model_svr.predict(x_train_ori)
pred_svr_train_score.append(r2_score(y_train_ori, pred_svr_ori_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.21832497545126242 0.24392381731385268今回は「0.21832」のスコアが基準となります。
そしてグラフ表示をしてみます。
<セル4>
from matplotlib import pyplot as plt
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
X軸が正解値、Y軸が予想値なので、予想精度が上がるにつれて右肩上がりのプロットになるはずです。
それでは始めていきましょう。
gamma
とは言っても共通のパラメータ以外で、rbfで影響のあるパラメータというのは「gamma」しかないようです。


ということでまずはこの「gamma」をいじってみましょう。
gamma: カーネルがrbf、poly、sigmoidの時のカーネル係数。
“scale”か”auto”かfloat値(小数)、デフォルト値は”scale”
ちなみに”scale”と”auto”の違いは、計算方法の違いのようです。
if
gamma='scale'(default) is passed then it uses 1 / (n_features * X.var()) as value of gamma,if ‘auto’, uses 1 / n_features.
https://scikit-learn.org/stable/modules/generated/sklearn.svm.SVR.html
“scale”がデフォルトということで、”auto”から試してみましょう。
<セル5 gamma="auto">
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="rbf", gamma="auto")
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
-0.03237276120034532 -0.0168603205685676一気にスコアが悪くなりました。
<セル6 gamma="auto">
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果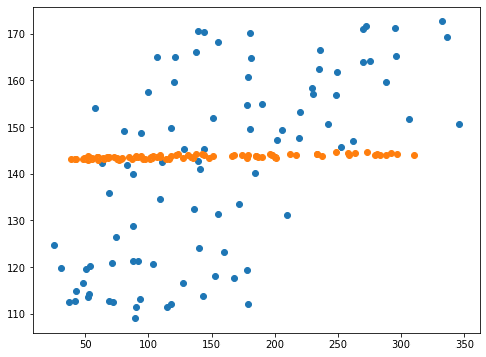
どうやら”auto”にすると、制限が強すぎて、予想値が一点に固まってしまうようです。
もちろんこれはデータによってかもしれません。
ただこのgammaのデフォルトがScikit-learnのバージョンが0.22から0.23にアップデートされた時に、”auto”から”scale”に変更になっています。
つまり一般的な状況において、”auto”よりも”scale”の方が良い結果をもたらすと考えていいのではないでしょうか。
次にgammaの値を変えて試していきましょう。
まずは負の数が使えるのか試してみましょう。
ということでgamma=-1としてみます。
<セル5 gamma=-1>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="rbf", gamma=-1)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
<ipython-input-15-dbd0abedb1ab> in <module>
7
8 model_svr = SVR(kernel="rbf", gamma=-1)
----> 9 model_svr.fit(x_train, y_train)
10 pred_svr = model_svr.predict(x_test)
11 pred_svr_score.append(r2_score(y_test, pred_svr))
/opt/anaconda3/lib/python3.7/site-packages/sklearn/svm/_base.py in fit(self, X, y, sample_weight)
215
216 seed = rnd.randint(np.iinfo('i').max)
--> 217 fit(X, y, sample_weight, solver_type, kernel, random_seed=seed)
218 # see comment on the other call to np.iinfo in this file
219
/opt/anaconda3/lib/python3.7/site-packages/sklearn/svm/_base.py in _dense_fit(self, X, y, sample_weight, solver_type, kernel, random_seed)
274 cache_size=self.cache_size, coef0=self.coef0,
275 gamma=self._gamma, epsilon=self.epsilon,
--> 276 max_iter=self.max_iter, random_seed=random_seed)
277
278 self._warn_from_fit_status()
sklearn/svm/_libsvm.pyx in sklearn.svm._libsvm.fit()
ValueError: gamma < 0エラーが出てしまいました。
エラーの最後の行の「gamma < 0」とあるように、gammaは正の数でないとダメなようです。
それでは正の数の小さい方から試していきます。
ということでgamma=0.1。
<セル5 gamma=0.1>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="rbf", gamma=0.1)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
-0.030478410546448585 -0.020573967712274883これまたかなり値が悪くなってしまっています。
グラフはどうでしょうか?
<セル6 gamma=0.1>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果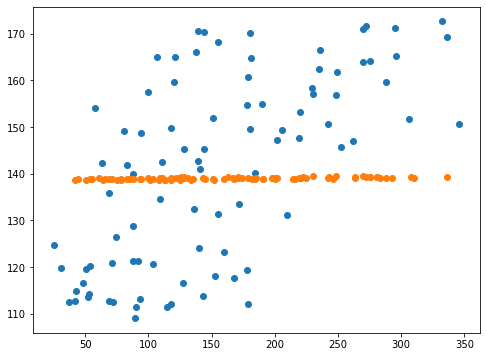
これまた一点に固まってしまっています。
これもまた係数による縛りが強すぎて、予想値が一点になってしまうというこれまでにもよくあった現象のようです。
ということはgammaの値を大きくすると縛りが弱くなっていくのでしょうか。
少し大きくしてみて、gamma=1を試してみましょう。
<セル5 gamma=1>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="rbf", gamma=1)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.006332032484584297 0.013291885167634087スコアは先ほどのgamma=0.1よりは改善しましたが、指定なしの「0.21832」と比べるとまだまだ低いです。
<セル6 gamma=1>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果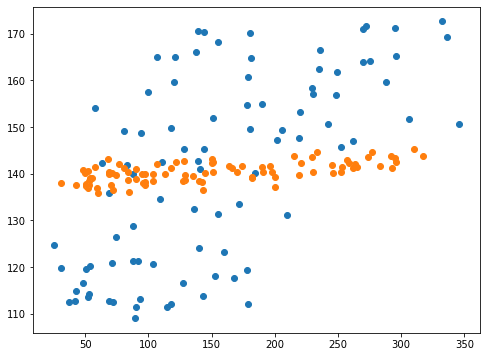
グラフを見てみると確かに予想値は広がってきています。
しかし基準のデータに比べるとまだまだ一点に固まっていますね。
ではgamma=10としてみましょう。
<セル5 gamma=10>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="rbf", gamma=10)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.1769332135544298 0.1906019684544688スコアとしてはだいぶ改善し、もう少しで指定なしと同じくらいになりそうです。
ただ目標としては指定なしよりも超えてくれるものを探しているので、まだまだ足りていません。
グラフの方はどうでしょうか。
<セル6 gamma=10>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果
先ほどよりもばらけていますが、それでもまだ何となく指定なし(青点)よりもばらつきが少ないように感じられます。
ということでgamma=100としてみましょう。
<セル5 gamma=100>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="rbf", gamma=100)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.21010999208529563 0.23330676541790007指定していない時のスコアが0.21832で、gamma=100のスコアが0.21011なので、これでスコア的には大体同じになりました。
<セル6 gamma=100>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果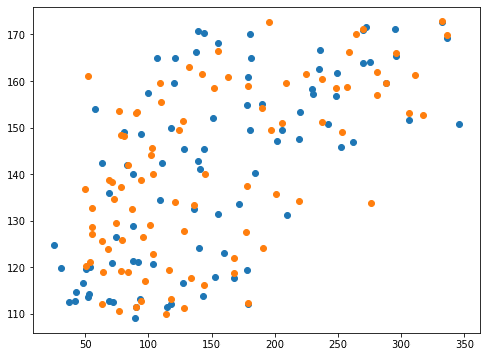
正解値vs予想値のプロットも指定なし(青点)とgamma=100(橙点)で同じくらいになりました。
ということはもっとgammaを大きくしたらさらに改善されるのでしょうか?
ではgamma=1000を試してみましょう。
<セル5 gamma=1000>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="rbf", gamma=1000)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
-0.02240310377589627 0.01571026418317856<セル6 gamma=1000>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果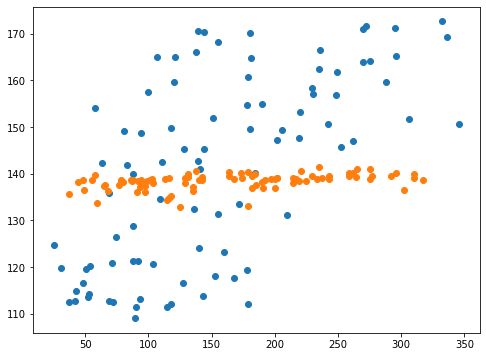
予想に反してスコアはかなり悪くなってしまい、予想値は一点に集中するようになってしまいました。
ということはgammaを大きくすると、予想値はばらけるというのは間違っていて、gammaをそのデータにとってちょうどいい値にすることで予想精度が良くなる。
ただしgammaの値がちょうどいい値から小さい方でも大きい方でも大きくずれていると予想値に縛りがかかり、予想値は一点に集中してしまう、と考えた方が良さそうです。
色々試してみたのですが、結局はやはりデフォルト値であるscaleを使うのが良さそうだなと感じました。
kernel=”linear”
今回はkernelのrbfだけにしようかと思ったのですが、コンテンツ量の関係上、kernel=”linear”とkernel=”precomputed”も試してみることにします。
ということでまずはkernel=”linear”から。
<セル7 kernel="linear">
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="linear")
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
-0.016747957553524807 -0.00449802976511355<セル8 kernel="linear">
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果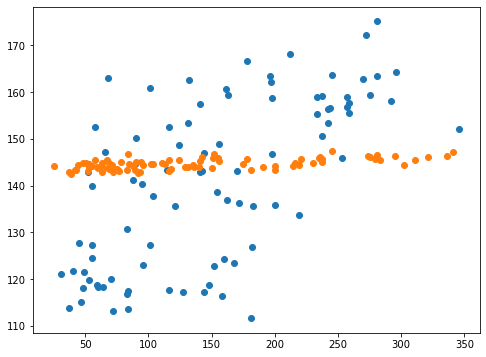
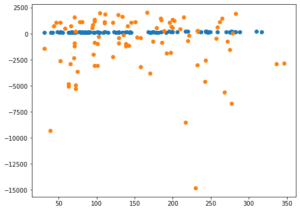
残念ながらkernel=”linear”はスコアがだいぶ悪くなってしまい、予想値も一点に集中してしまいました。
私の予想では、SVRはサポートベクター回帰というように、SVM(サポートベクターマシン)のように、グラフ上に線を引いて、その線をもとに回帰を行うのだと考えられます。
そしてkernelによりその線の引き方を決めるのですが、linearということは直線で線を引くということ。
つまり線の引き方に制限が大きい、引き方なわけです。
そのため、kernel=”linear”では予想精度であるスコアが悪くなってしまったのだと考えられます。
kernel=”precomputed”
次はkernel=”precomputed”を試してみましょう。
正直これは何のことだかよく分からないので、まずは試してみるのが早いかなという判断です。
<セル9 kernel="precomputed">
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(kernel="precomputed")
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
<ipython-input-20-e9346c6cd4e7> in <module>
7
8 model_svr = SVR(kernel="precomputed")
----> 9 model_svr.fit(x_train, y_train)
10 pred_svr = model_svr.predict(x_test)
11 pred_svr_score.append(r2_score(y_test, pred_svr))
/opt/anaconda3/lib/python3.7/site-packages/sklearn/svm/_base.py in fit(self, X, y, sample_weight)
179 raise ValueError("Precomputed matrix must be a square matrix."
180 " Input is a {}x{} matrix."
--> 181 .format(X.shape[0], X.shape[1]))
182
183 if sample_weight.shape[0] > 0 and sample_weight.shape[0] != n_samples:
ValueError: Precomputed matrix must be a square matrix. Input is a 353x5 matrix.エラーが出てしまいました。
最後の一文からすると何かのマトリックス(行列)が必要なようです。
そこで一度解説に戻ってみましょう。
kernel{‘linear’, ‘poly’, ‘rbf’, ‘sigmoid’, ‘precomputed’}, default=’rbf’
Specifies the kernel type to be used in the algorithm. It must be one of ‘linear’, ‘poly’, ‘rbf’, ‘sigmoid’, ‘precomputed’ or a callable. If none is given, ‘rbf’ will be used. If a callable is given it is used to precompute the kernel matrix.
https://scikit-learn.org/stable/modules/generated/sklearn.svm.SVR.html
最後の「it is used to precompute the kernel matrix」から推測するにprecomputedではそれぞれのデータに対してのkernelのマトリックスが必要になるようです。
つまりデータ毎に異なる処理をする時に使うのだと考えられます。
とりあえず通常は使わなさそうなので、今回はここまでとしましょう。
あと残るは”poly”と”sigmoid”です。
次回は”poly”のオプションを試していくことにしましょう。
ということで今回はこんな感じで。

コメント