機械学習ライブラリScikit-learn
前回、機械学習ライブラリScikit-learnの糖尿病患者のデータセットを使い、RidgeRegressionモデルのオプションを試してみました。
今回はSVRモデルのオプションを試していきましょう。
ということでいつも通りデータの読み込みから。
<セル1>
from sklearn.datasets import load_diabetes
import pandas as pd
diabetes = load_diabetes()
df = pd.DataFrame(diabetes.data, columns=diabetes.feature_names)
df["target"] = diabetes.target
df
実行結果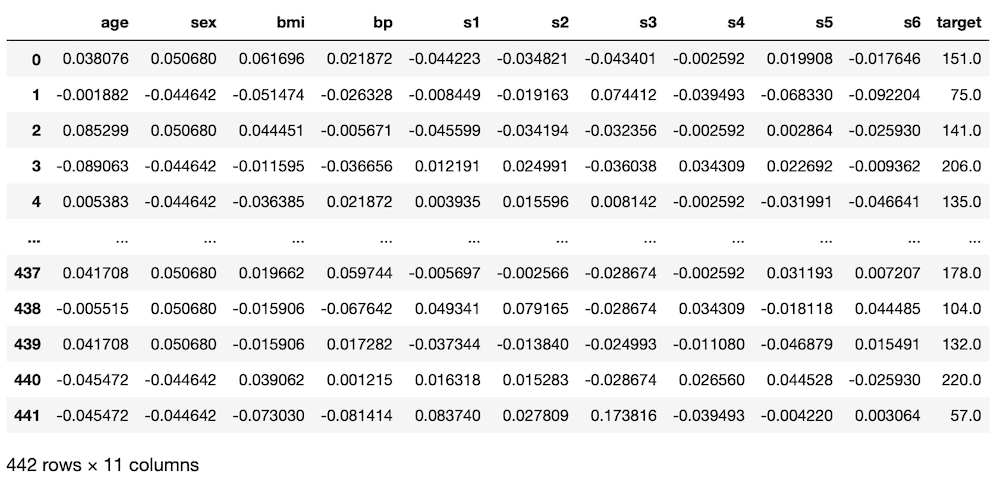
次に機械学習に用いる特徴量とターゲットをそれぞれ変数xとyに格納します。
(後で気付いたのですが、前の記事では「s6」も関連性がありそうということでしたが、忘れてしまいました。他の記事でも上記の組み合わせで行っているものもありますが、忘れたんだなぁと思って読んでください。)
<セル2>
x = df.loc[:, ["bmi", "s5", "bp", "s4", "s3"]]
y = df.loc[:, "target"]
実行結果ではSVRモデルを読み込んで、機械学習と予想を100回試した後、その予想精度のスコアの平均値を表示します。
その際、過学習になっていないか確認するため、テスト用データでのスコアだけでなく、学習用データでのスコアも計算します。
<セル3>
from sklearn.model_selection import train_test_split
from sklearn.svm import SVR
from sklearn.metrics import r2_score
import numpy as np
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train_ori, x_test_ori, y_train_ori, y_test_ori = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR()
model_svr.fit(x_train_ori, y_train_ori)
pred_svr_ori = model_svr.predict(x_test_ori)
pred_svr_score.append(r2_score(y_test_ori, pred_svr_ori))
pred_svr_ori_train = model_svr.predict(x_train_ori)
pred_svr_train_score.append(r2_score(y_train_ori, pred_svr_ori_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.22315444797654346 0.2444680512648118SVRのスコアは結構低めの0.22315と出てきました。
とりあえず最後の学習で得られたデータをグラフにしてみます。
<セル4>
from matplotlib import pyplot as plt
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
実行結果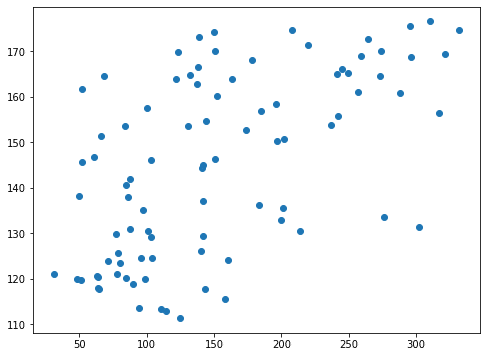
なんというかバラバラという感じで一貫性があるようには見えませんね。
それではオプションをいじることでこれがどう変わっていくのか、楽しみながいじっていきましょう。
SVRのヘルプ
ここもいつも通り先にヘルプを見て、どんなオプションがあるのか確認していきましょう。
<セル5>
print(help(SVR))
実行結果
Help on class SVR in module sklearn.svm._classes:
class SVR(sklearn.base.RegressorMixin, sklearn.svm._base.BaseLibSVM)
| SVR(*, kernel='rbf', degree=3, gamma='scale', coef0=0.0, tol=0.001, C=1.0, epsilon=0.1, shrinking=True, cache_size=200, verbose=False, max_iter=-1)
|
| Epsilon-Support Vector Regression.
|
| The free parameters in the model are C and epsilon.
|
| The implementation is based on libsvm. The fit time complexity
| is more than quadratic with the number of samples which makes it hard
| to scale to datasets with more than a couple of 10000 samples. For large
| datasets consider using :class:`sklearn.svm.LinearSVR` or
| :class:`sklearn.linear_model.SGDRegressor` instead, possibly after a
| :class:`sklearn.kernel_approximation.Nystroem` transformer.
|
| Read more in the :ref:`User Guide <svm_regression>`.
|
| Parameters
| ----------
| kernel : {'linear', 'poly', 'rbf', 'sigmoid', 'precomputed'}, default='rbf'
| Specifies the kernel type to be used in the algorithm.
| It must be one of 'linear', 'poly', 'rbf', 'sigmoid', 'precomputed' or
| a callable.
| If none is given, 'rbf' will be used. If a callable is given it is
| used to precompute the kernel matrix.
|
| degree : int, default=3
| Degree of the polynomial kernel function ('poly').
| Ignored by all other kernels.
|
| gamma : {'scale', 'auto'} or float, default='scale'
| Kernel coefficient for 'rbf', 'poly' and 'sigmoid'.
|
| - if ``gamma='scale'`` (default) is passed then it uses
| 1 / (n_features * X.var()) as value of gamma,
| - if 'auto', uses 1 / n_features.
|
| .. versionchanged:: 0.22
| The default value of ``gamma`` changed from 'auto' to 'scale'.
|
| coef0 : float, default=0.0
| Independent term in kernel function.
| It is only significant in 'poly' and 'sigmoid'.
|
| tol : float, default=1e-3
| Tolerance for stopping criterion.
|
| C : float, default=1.0
| Regularization parameter. The strength of the regularization is
| inversely proportional to C. Must be strictly positive.
| The penalty is a squared l2 penalty.
|
| epsilon : float, default=0.1
| Epsilon in the epsilon-SVR model. It specifies the epsilon-tube
| within which no penalty is associated in the training loss function
| with points predicted within a distance epsilon from the actual
| value.
|
| shrinking : bool, default=True
| Whether to use the shrinking heuristic.
| See the :ref:`User Guide <shrinking_svm>`.
|
| cache_size : float, default=200
| Specify the size of the kernel cache (in MB).
|
| verbose : bool, default=False
| Enable verbose output. Note that this setting takes advantage of a
| per-process runtime setting in libsvm that, if enabled, may not work
| properly in a multithreaded context.
|
| max_iter : int, default=-1
| Hard limit on iterations within solver, or -1 for no limit.今回はなかなか分量が多そうです。
そして何より、オプションで選択したものに対して使用できる、もしくは効果的なオプションがあるということでなかなか複雑そうです。
その中でも特に重要そうなのが「kernel」です。
kernel: SVRはSVMと同様にデータをプロットしたグラフに対して、特定の線を引き、プロットを分類していく。その際にどのように線を引くのかをkernelのオプションで設定する。
選択肢は「linear」、「poly」、「rbf」、「sigmoid」、「precomputed」の5つ。
デフォルト値は「rbf」
今回はこのkernelはデフォルト値(rbf)のままにしておいて、他のオプションの中で特定のものだけに有効というものでは”ない”ものを試していきましょう。
「tol」に関してはLassoモデルの時に解説しているので、「C」、「epsilon」、「shrinking」を試してみましょう。
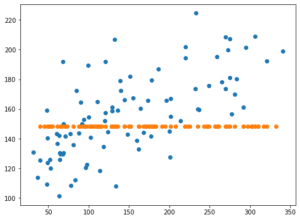
C
C: 正則化のパラメータ。正則化の強度はCに反比例する。Cは正の値である必要がある。
float値(小数)、デフォルト値は1.0
つまりCが小さくなればなるほど強く正則化され、大きくなればなるほど正則化が弱くなるということです。
ということでCを0.1と10にして試してみましょう。
<セル5 C=0.1>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(C=0.1)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.005947933620048027 0.016379096095077424<セル7 C=0.1>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果
C=0.1では正則化が強くなり過ぎて、予想値が一点に集まってきてしまいました。
それに伴いスコアも激減しています。
次にC=10を試してみましょう。
<セル6 C=10>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(C=10)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.46265714876109454 0.4933511689453928<セル7 C=10>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果
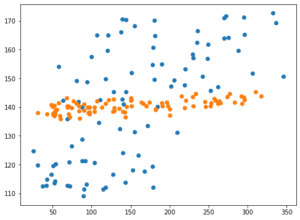
C=10の場合は正則化が弱くなり、全体的に値がばらけました。
それでもX軸に正解値、Y軸に予想値をとるとちょっと右肩上がりのグラフになり、傾向は掴めている感じがします。
epsilon
epsilon: epsilon-SVRモデルで用いるパラメータ。
float値(小数)、デフォルト値は1.0
このパラメータを使うとeplison-SVRモデルとなるようだが、詳しいことは不明。
そういう場合はやってみるに限ります。
特に値の制限は書いてなかったので、マイナスでも行けるんでしょうか?
まずはepsilon=-10としてみます。
<セル8 epsilon=-10>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(epsilon=-10)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
<ipython-input-22-641ee0335d62> in <module>
7
8 model_svr = SVR(epsilon=-10)
----> 9 model_svr.fit(x_train, y_train)
10 pred_svr = model_svr.predict(x_test)
11 pred_svr_score.append(r2_score(y_test, pred_svr))
/opt/anaconda3/lib/python3.7/site-packages/sklearn/svm/_base.py in fit(self, X, y, sample_weight)
215
216 seed = rnd.randint(np.iinfo('i').max)
--> 217 fit(X, y, sample_weight, solver_type, kernel, random_seed=seed)
218 # see comment on the other call to np.iinfo in this file
219
/opt/anaconda3/lib/python3.7/site-packages/sklearn/svm/_base.py in _dense_fit(self, X, y, sample_weight, solver_type, kernel, random_seed)
274 cache_size=self.cache_size, coef0=self.coef0,
275 gamma=self._gamma, epsilon=self.epsilon,
--> 276 max_iter=self.max_iter, random_seed=random_seed)
277
278 self._warn_from_fit_status()
sklearn/svm/_libsvm.pyx in sklearn.svm._libsvm.fit()
ValueError: epsilon < 0エラーになりました。
どうやらepsilonは正の値でないとダメなようです。
それではepsilon=0.1と10を試してみましょう。
<セル8 epsilon=0.1>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(epsilon=0.1)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.22675345376178913 0.2441227541474042<セル9 epsilon=0.1>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果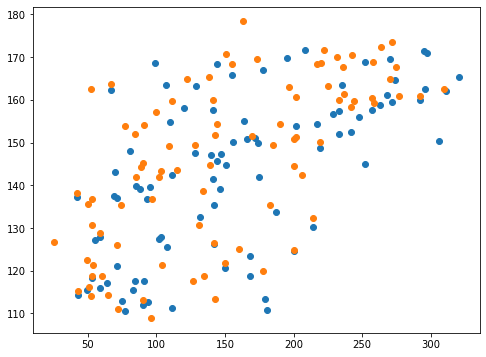
あまり変わったようには見受けられませんね。
次にepsilon=10を試してみましょう。
<セル8 epsilon=10>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(epsilon=10)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.2138257287042894 0.2411703560729935<セル9 epsilon=10>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果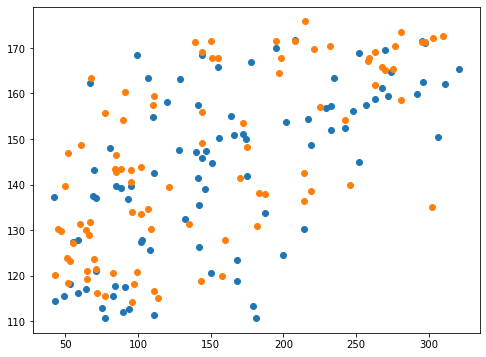
こちらも変わったように見えません。
残念ながらよく分からないオプションでした。
shrinking
shrinking: 機械学習の時に重要でない特徴量の寄与率を下げて計算する。
bool値(True or False)、デフォルト値はTrue
このShrinkingは特徴量の重みを変動させるパラメータのようです。
つまりTrueにすると重要な特徴量は学習により影響するようになり、逆に重要でない特徴量の影響は少なくなるということです。
となるとFalseにすると使用する特徴量全てが同等に扱われるということでしょうか。
それならば予想精度に大きく影響を与える可能性があるのではないでしょうか。
ということで試してみましょう。
<セル10 shrinking=False>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(shrinking=False)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.22462490074150857 0.24478445432761942<セル11 shrinking=True>
fig=plt.figure(figsize=(8,6))
plt.clf()
plt.scatter(y_test_ori, pred_svr_ori)
plt.scatter(y_test, pred_svr)
実行結果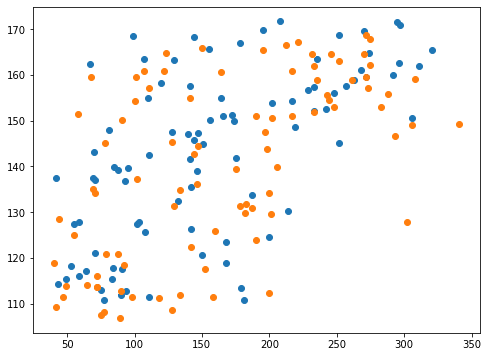
結果に大きく影響するのではないかと予想したのですが、思ったよりも影響が見られませんでした。
となると最初に指定した特徴量が少ないため、寄与率の変化がなかったことが考えられます。
ということでデータセットにある全ての特徴量を使って試してみましょう。
<セル12>
x = df.loc[:, ["age", "sex", "bmi", "bp", "s1", "s2", "s3", "s4", "s5", "s6"]]
y = df.loc[:, "target"]
実行結果まずはShrinkingを指定せずに試してみます。
デフォルト値がTrueなので、結果はshrinking=Trueの結果になります。
<セル13 Shrinking指定なし(True)>
from sklearn.model_selection import train_test_split
from sklearn.svm import SVR
from sklearn.metrics import r2_score
import numpy as np
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train_ori, x_test_ori, y_train_ori, y_test_ori = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR()
model_svr.fit(x_train_ori, y_train_ori)
pred_svr_ori = model_svr.predict(x_test_ori)
pred_svr_score.append(r2_score(y_test_ori, pred_svr_ori))
pred_svr_ori_train = model_svr.predict(x_train_ori)
pred_svr_train_score.append(r2_score(y_train_ori, pred_svr_ori_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.16340145098760794 0.1780150813150479次にShrinkingをFalseにして試してみます。
<セル13 Shrinking=False>
trial = 100
pred_svr_score = []; pred_svr_train_score = []
for i in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR(shrinking=False)
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_train = model_svr.predict(x_train)
pred_svr_train_score.append(r2_score(y_train, pred_svr_train))
pred_svr_ave = np.average(np.array(pred_svr_score))
pred_svr_train_ave = np.average(np.array(pred_svr_train_score))
print(pred_svr_ave, pred_svr_train_ave)
実行結果
0.1550248208498759 0.17806739218503403“shrinking=True”で「0.16340」、”shrinking=False”で「0.15502」ということなので、shrinkingのあるなしで特に大きな違いはないように見受けられます。
ということでshrinkingはまずはデフォルトのTrueで問題ないのではないでしょうか。
次回はSVRのkernelのうちデフォルトのrbf特有のオプションを見ていきましょう。
ではでは今回はこんな感じで。

コメント