機械学習ライブラリScikit-learn
前回、機械学習ライブラリScikit-learnの糖尿病患者のデータセットを使い、特徴量の全ての組み合わせを試すプログラムを作成しました。
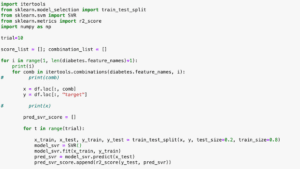
今回はそのプログラムを使って、SVRモデルで特徴量の全ての組み合わせを試し、どんな特徴量の組み合わせを使うのがいいのか検討してみたいと思います。
まずは前回のプログラムのおさらいから。
<セル1>
from sklearn.datasets import load_diabetes
import pandas as pd
diabetes = load_diabetes()
df = pd.DataFrame(diabetes.data, columns=diabetes.feature_names)
df["target"] = diabetes.target
df
実行結果
<セル2>
import itertools
from sklearn.model_selection import train_test_split
from sklearn.svm import SVR
from sklearn.metrics import r2_score
import numpy as np
trial=10
score_list = []; combination_list = []
for i in range(1, len(diabetes.feature_names)+1):
print(i)
for comb in itertools.combinations(diabetes.feature_names, i):
# print(comb)
x = df.loc[:, comb]
y = df.loc[:, "target"]
# print(x)
pred_svr_score = []
for t in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
model_svr = SVR()
model_svr.fit(x_train, y_train)
pred_svr = model_svr.predict(x_test)
pred_svr_score.append(r2_score(y_test, pred_svr))
pred_svr_ave = np.average(np.array(pred_svr_score))
# print(pred_svr_ave)
if len(score_list) < 10:
score_list.append(pred_svr_ave)
combination_list.append(comb)
elif len(score_list) == 10:
if np.min(score_list) < pred_svr_ave:
del combination_list[np.argmin(score_list)]
del score_list[np.argmin(score_list)]
combination_list.append(comb)
score_list.append(pred_svr_ave)
for comb, score in zip(combination_list, score_list):
print(comb, score)
実行結果
1
2
3
4
5
6
7
8
9
10
('s5',) 0.2324347008540927
('bmi', 's5') 0.25211732190510483
('bmi', 'bp', 's5') 0.2590815528704341
('bmi', 's2', 's5') 0.24376496089529925
('bmi', 'bp', 's2', 's5') 0.24234159140148792
('bmi', 'bp', 's3', 's5') 0.2270951116349605
('bmi', 'bp', 's4', 's5') 0.22518801559600696
('bmi', 's4', 's5', 's6') 0.2313280622825627
('bmi', 'bp', 's3', 's4', 's5') 0.24303507844402308
('bmi', 'bp', 's2', 's3', 's4', 's5') 0.22608041336148746今回はこのプログラムを5回実行してみて、TOP10のスコアに入った特徴量をカウントしていくことにしましょう。
プログラムを5回実行してみた
1回目の実行結果
| 特徴量 | スコア | 順位 |
| ‘s5’ | 0.23243 | 6 |
| ‘bmi’, ‘s5’ | 0.25211 | 2 |
| ‘bmi’, ‘bp’, ‘s5’ | 0.25908 | 1 |
| ‘bmi’, ‘s2’, ‘s5’ | 0.24376 | 3 |
| ‘bmi’, ‘bp’, ‘s2’, ‘s5’ | 0.24234 | 5 |
| ‘bmi’, ‘bp’, ‘s3’, ‘s5’ | 0.22710 | 8 |
| ‘bmi’, ‘bp’, ‘s4’, ‘s5’ | 0.22519 | 10 |
| ‘bmi’, ‘s4’, ‘s5’, ‘s6’ | 0.23133 | 7 |
| ‘bmi’, ‘bp’, ‘s3’, ‘s4’, ‘s5’ | 0.24304 | 4 |
| ‘bmi’, ‘bp’, ‘s2’, ‘s3’, ‘s4’, ‘s5’ | 0.22608 | 9 |
| age | sex | bmi | bp | s1 | s2 | s3 | s4 | s5 | s6 |
| 0 | 0 | 9 | 6 | 0 | 3 | 3 | 4 | 10 | 1 |
第一位:’bmi’, ‘bp’, ‘s5’
2回目の実行結果
| 特徴量 | スコア | 順位 |
| ‘bmi’, ‘s5’ | 0.28445 | 1 |
| ‘age’, ‘bmi’, ‘s5’ | 0.23399 | 8 |
| ‘bmi’, ‘bp’, ‘s5’ | 0.23702 | 7 |
| ‘bmi’, ‘s3’, ‘s5’ | 0.24538 | 4 |
| ‘bmi’, ‘s4’, ‘s5’ | 0.25168 | 2 |
| ‘age’, ‘bmi’, ‘s4’, ‘s5’ | 0.23244 | 10 |
| ‘bmi’, ‘bp’, ‘s4’, ‘s5’ | 0.24912 | 3 |
| ‘bmi’, ‘s3’, ‘s5’, ‘s6’ | 0.24316 | 5 |
| ‘age’, ‘bmi’, ‘bp’, ‘s4’, ‘s5’ | 0.23350 | 9 |
| ‘bmi’, ‘bp’, ‘s3’, ‘s5’, ‘s6’ | 0.24127 | 6 |
| age | sex | bmi | bp | s1 | s2 | s3 | s4 | s5 | s6 |
| 3 | 0 | 10 | 4 | 0 | 0 | 3 | 4 | 10 | 2 |
第一位:’bmi’, ‘s5’
3回目の実行結果
| 特徴量 | スコア | 順位 |
| ‘s5’ | 0.24978 | 2 |
| ‘bmi’, ‘s5’ | 0.25060 | 1 |
| ‘bmi’, ‘bp’, ‘s5’ | 0.24121 | 4 |
| ‘bmi’, ‘s1’, ‘s5’ | 0.23656 | 6 |
| ‘bmi’, ‘s4’, ‘s5’ | 0.24570 | 3 |
| ‘bmi’, ‘bp’, ‘s2’, ‘s5’ | 0.22855 | 10 |
| ‘bmi’, ‘bp’, ‘s5’, ‘s6’ | 0.22878 | 9 |
| ‘bmi’, ‘bp’, ‘s2’, ‘s3’, ‘s5’ | 0.23071 | 7 |
| ‘bmi’, ‘bp’, ‘s3’, ‘s4’, ‘s5’ | 0.23880 | 5 |
| ‘bmi’, ‘s2’, ‘s3’, ‘s4’, ‘s5’ | 0.23023 | 8 |
| age | sex | bmi | bp | s1 | s2 | s3 | s4 | s5 | s6 |
| 0 | 0 | 9 | 5 | 1 | 3 | 3 | 3 | 10 | 1 |
第一位:’bmi’, ‘s5’
4回目の実行結果
| 特徴量 | スコア | 順位 |
| ‘s5’ | 0.25121 | 3 |
| ‘bmi’, ‘s5’ | 0.27629 | 1 |
| ‘s3’, ‘s5’ | 0.24068 | 4 |
| ‘bmi’, ‘bp’, ‘s5’ | 0.26066 | 2 |
| ‘bmi’, ‘s1’, ‘s5’ | 0.23717 | 6 |
| ‘bmi’, ‘s5’, ‘s6’ | 0.23921 | 5 |
| ‘age’, ‘bmi’, ‘bp’, ‘s5’ | 0.23365 | 9 |
| ‘age’, ‘bmi’, ‘s4’, ‘s5’ | 0.23215 | 10 |
| ‘bmi’, ‘bp’, ‘s4’, ‘s5’ | 0.23644 | 7 |
| ‘bmi’, ‘bp’, ‘s3’, ‘s4’, ‘s5’ | 0.23526 | 8 |
| age | sex | bmi | bp | s1 | s2 | s3 | s4 | s5 | s6 |
| 2 | 0 | 8 | 4 | 1 | 0 | 2 | 3 | 10 | 1 |
第一位:’bmi’, ‘s5’
5回目の実行結果
| 特徴量 | スコア | 順位 |
| ‘bmi’, ‘s5’ | 0.27083 | 1 |
| ‘bp’, ‘s5’ | 0.23551 | 6 |
| ‘bmi’, ‘bp’, ‘s5’ | 0.24507 | 3 |
| ‘age’, ‘bmi’, ‘s2’, ‘s5’ | 0.23561 | 5 |
| ‘bmi’, ‘bp’, ‘s3’, ‘s5’ | 0.24666 | 2 |
| ‘bmi’, ‘bp’, ‘s4’, ‘s5’ | 0.23516 | 7 |
| ‘bmi’, ‘bp’, ‘s1’, ‘s4’, ‘s5’ | 0.23184 | 10 |
| ‘bmi’, ‘bp’, ‘s2’, ‘s4’, ‘s5’ | 0.23434 | 8 |
| ‘bmi’, ‘bp’, ‘s3’, ‘s4’, ‘s5’ | 0.23753 | 4 |
| ‘bmi’, ‘bp’, ‘s3’, ‘s5’, ‘s6’ | 0.23235 | 9 |
| age | sex | bmi | bp | s1 | s2 | s3 | s4 | s5 | s6 |
| 1 | 0 | 9 | 8 | 1 | 2 | 3 | 4 | 10 | 1 |
第一位:’bmi’, ‘s5’
各特徴量の出現回数まとめ
| age | sex | bmi | bp | s1 | s2 | s3 | s4 | s5 | s6 |
| 6 | 0 | 45 | 27 | 3 | 8 | 14 | 18 | 50 | 6 |
第一位の組み合わせで一番多かったもの:’bmi’, ‘s5’
こうみると「s5」、「bmi」は文句なく学習させる特徴量として使うべきでしょう。
前に相関マップを表示し、選択した特徴量は「bmi」、「s5」、「bp」、「s4」、「s3」、「s6」でした。
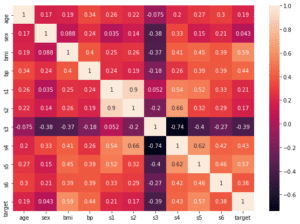
今回の結果から言うと、「bp」、「s3」、「s4」は他の特徴量よりも出現回数が有意に多いことから、確かに選択してもいい特徴量だと考えられます。
ただし「s6」の出現回数は「age」や「s2」の出現回数と変わらず、有意に多いとは言えなさそうです。
結論としては、SVRモデルでは「s5」、「bmi」を特徴量として学習させれば十分ということです。
ただ「bp」、「s4」、「s3」もこの順を優先度として加えてもいいかもしれないです。
ここで一つ疑問が湧いてきました。
多分、どの機械学習モデルを使っても、「s5」、「bmi」、そして「bp」くらいまでは特徴量として重要だと言う結論が導き出されるでしょう。
では他の特徴量の影響は、機械学習モデルによって違うのでしょうか?
ということで次回はSVRモデルに替えて、RidgeRegressionモデルを使って同じ検討をしてみましょう。

ではでは今回はこんな感じで。

コメント