機械学習ライブラリScikit-learn
前回、機械学習ライブラリScikit-learnで複数の機械学習モデルで特徴量全ての組み合わせを試し、RidgeRegressionモデルに対して使われやすい特徴量をグラフ化するプログラムを作成しました。
今回はそのプログラムを使って分析を進めていきたいと思います。
まずは前回のおさらいから。
<セル1>
from sklearn.datasets import load_diabetes
import pandas as pd
diabetes = load_diabetes()
df = pd.DataFrame(diabetes.data, columns=diabetes.feature_names)
df["target"] = diabetes.target
df
実行結果
<セル2>
import itertools
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LinearRegression
from sklearn.linear_model import Lasso
from sklearn.linear_model import ElasticNet
from sklearn.linear_model import Ridge
from sklearn.svm import SVR
from sklearn.metrics import r2_score
import numpy as np
models = ["LinearRegression","Lasso", "ElasticNet", "RidgeRegression", "SVR"]
trial=10
score_list = []; combination_list = []; model_list = []
std_list = []; max_list = []; min_list = []; score_std_list = []
for i in range(1, len(diabetes.feature_names)+1):
print(i)
for comb in itertools.combinations(diabetes.feature_names, i):
# print(comb)
x = df.loc[:, comb]
y = df.loc[:, "target"]
# print(x)
for mod in models:
pred_score = []
for t in range(trial):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.2, train_size=0.8)
if mod == "LinearRegression":
model = LinearRegression()
elif mod == "Lasso":
model = Lasso()
elif mod == "ElasticNet":
model = ElasticNet()
elif mod == "RidgeRegression":
model = Ridge()
elif mod == "SVR":
model = SVR()
model.fit(x_train, y_train)
pred = model.predict(x_test)
pred_score.append(r2_score(y_test, pred))
# print(mod, r2_score(y_test, pred))
combination_list.append(comb)
score_list.append(np.average(np.array(pred_score)))
std_list.append(np.std(np.array(pred_score)))
max_list.append(np.max(np.array(pred_score)))
min_list.append(np.min(np.array(pred_score)))
model_list.append(mod)
results = pd.DataFrame()
results["Model_name"] = model_list
results["Combination"] = combination_list
results["Score"] = score_list
results["Max"] = max_list
results["Min"] = min_list
results["STD"] = std_list
results["Score/STD"] = results["Score"]/results["STD"]
results = results.sort_values("Score/STD", ascending=False)
results.head(10)
実行結果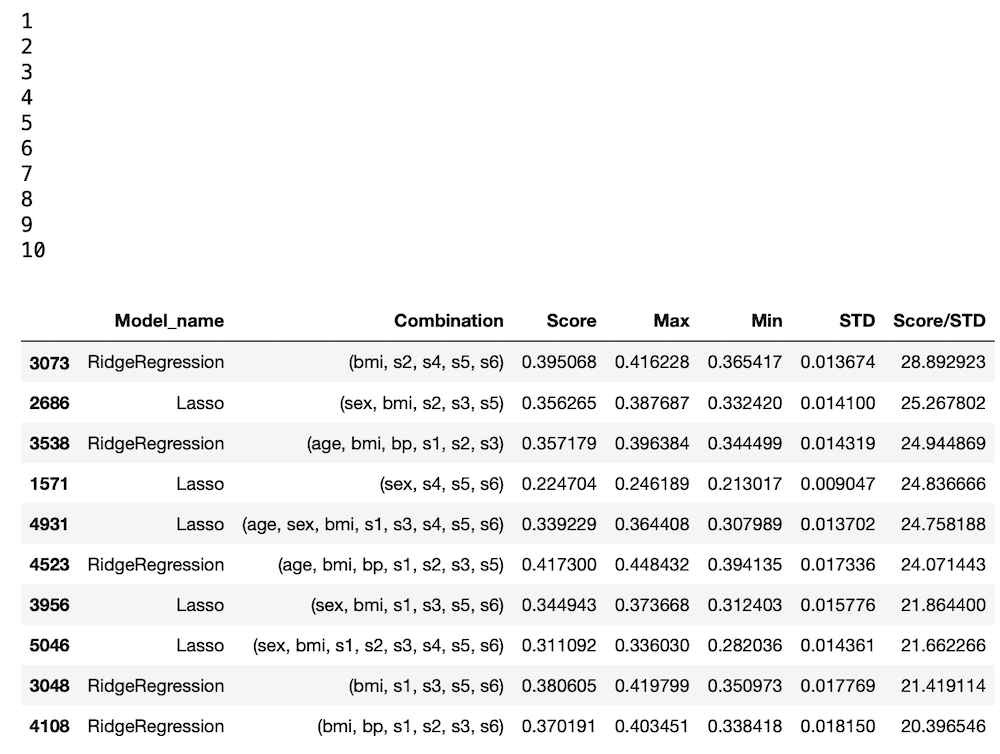
<セル3>
comb_count = []
for i in range(len(results)):
comb_count.append(len(results.iloc[i]["Combination"]))
results["No_comb"] = comb_count
results
実行結果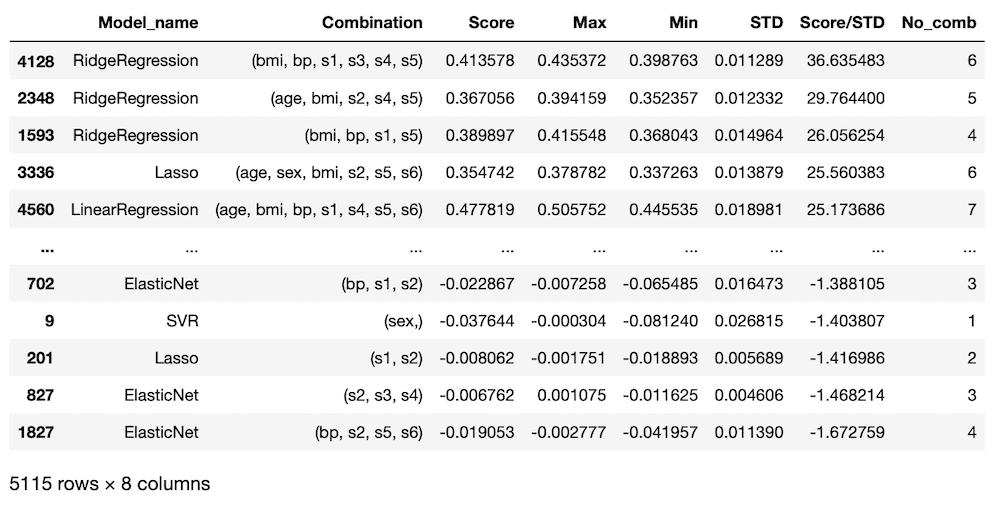
<セル4>
rd_data = results[results["Model_name"] == "RidgeRegression"]
rd_data
実行結果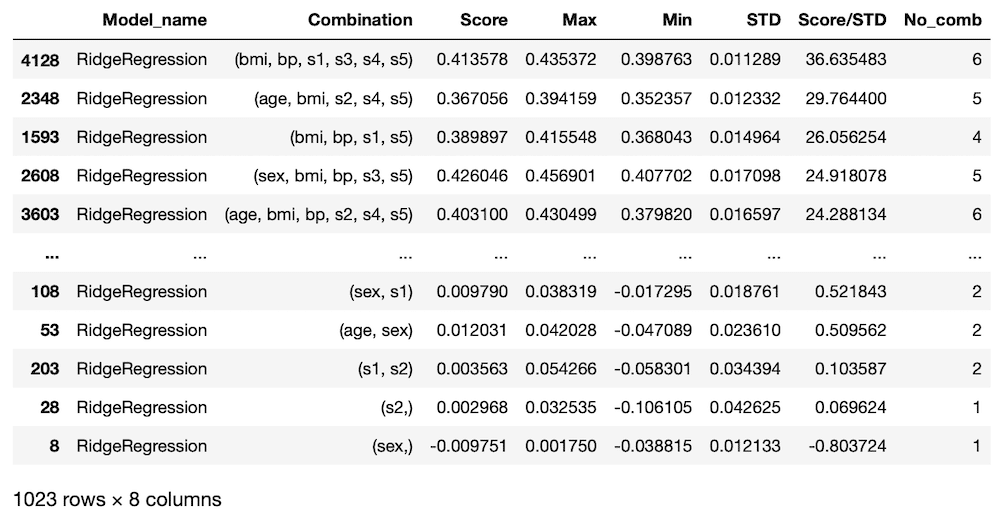
<セル5>
ranking = 100
rd_ranking = rd_data[:ranking]
age_count = rd_ranking["Combination"].sum().count("age")
sex_count = rd_ranking["Combination"].sum().count("sex")
bmi_count = rd_ranking["Combination"].sum().count("bmi")
bp_count = rd_ranking["Combination"].sum().count("bp")
s1_count = rd_ranking["Combination"].sum().count("s1")
s2_count = rd_ranking["Combination"].sum().count("s2")
s3_count = rd_ranking["Combination"].sum().count("s3")
s4_count = rd_ranking["Combination"].sum().count("s4")
s5_count = rd_ranking["Combination"].sum().count("s5")
s6_count = rd_ranking["Combination"].sum().count("s6")
names = ["age", "sex", "bmi", "bp", "s1", "s2", "s3", "s4", "s5", "s6"]
counts = [age_count,sex_count, bmi_count, bp_count, s1_count, s2_count, s3_count, s4_count, s5_count, s6_count]
print(names)
print(counts)
実行結果
['age', 'sex', 'bmi', 'bp', 's1', 's2', 's3', 's4', 's5', 's6']
[46, 48, 82, 60, 51, 52, 55, 45, 78, 49]<セル6>
from matplotlib import pyplot as plt
%matplotlib inline
fig = plt.figure(figsize=(8,6))
plt.clf()
plt.bar(names, counts)
実行結果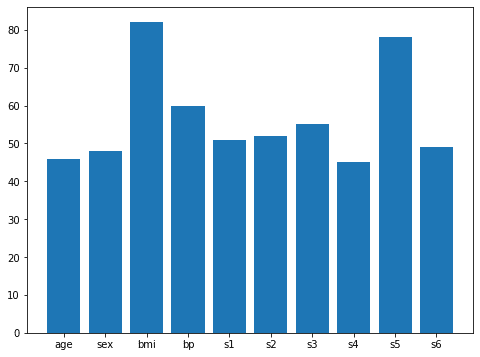
<セル7>
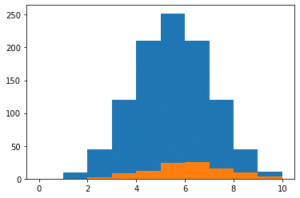
plt.hist(rd_data["No_comb"],range=(0, 10))
plt.hist(rd_ranking["No_comb"], range=(0,10))
実行結果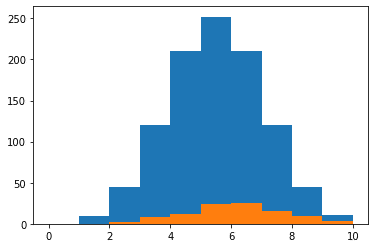
ということで分析していきましょう。
5回試行してみる
1回目
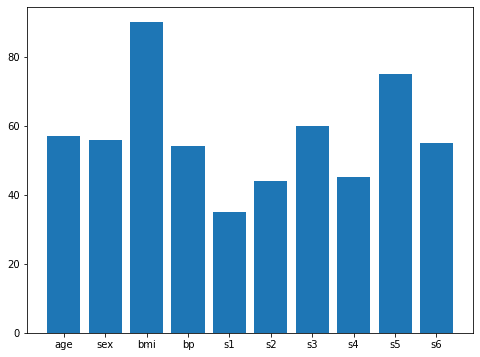
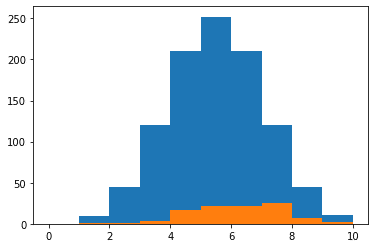
使用される特徴量の順はbmi、s5、s3で、個数は7個が最大。
2回目
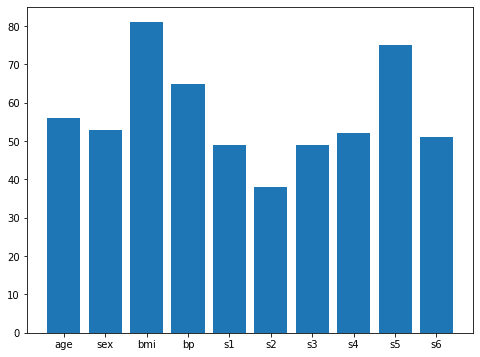
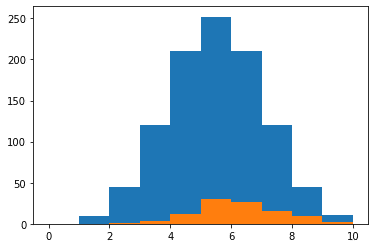
使用される特徴量の順はbmi、s5、bpで、個数は5個が最大。
3回目
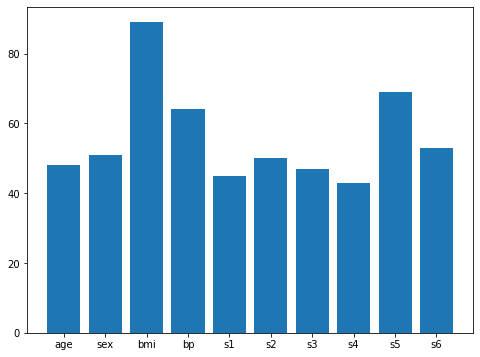
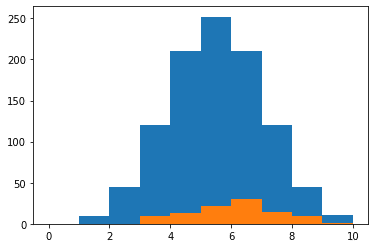
使用される特徴量の順はbmi、s5、bpで、個数は6個が最大。
4回目
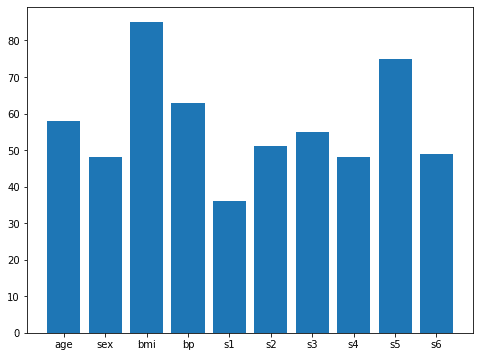
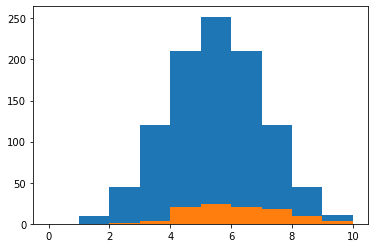
使用される特徴量の順はbmi、s5、bpで、個数は5個が最大。
5回目
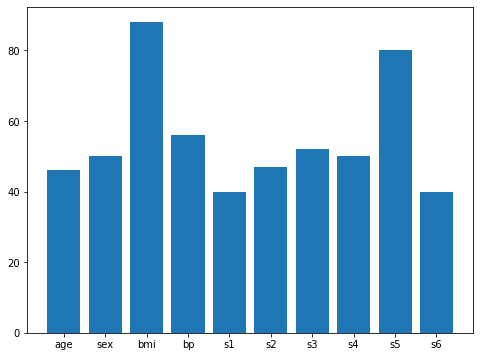
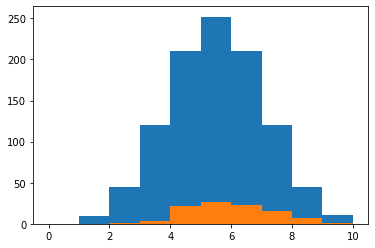
使用される特徴量の順はbmi、s5、bpで、個数は5個が最大。
分析まとめ
ということで5回プログラムを回してみました。
その結果、よく使われる特徴量としてはやはりbmiが一番多く、次にs5。
あとはbpがその後に続くといった感じでしょうか。
一度、s3が3位に来ていましたが、他の回では落ち込んでいることもあり、それほど重要そうな特徴量には見えません。
ちなみに最初に相関マップを作成し、分析した際は、
ということでじっと見ていくと、どうやら「bmi」、「s5」、「bp」、「s4」、「s3」、「s6」あたりが相関がありそうです。
https://3pysci.com/python-sklearn-19/
といっているので、結果が大きく変わったということではないですね。
ということはやはり相関マップを元に使用する特徴量を決めればよさそうです。
また使われる特徴量の個数は5個か6個ということが多かったのですが、ここら辺は組み合わせの数自体が多いところなので、選ばれやすい可能性が高いです。
また使われた特徴量の個数を組み合わせの個数で割り算した値を評価値としようともしたのですが、この場合、組み合わせ数が少ないものの方が良い評価値になりやすいのでやめました。
となるとせっかく分析した使われる特徴量の個数ですが、ちょっと使えない指標だったかもしれません。
ということで次は何をしようと考えたのですが、特徴量を詳しくみてみると、
「Attribute Information:」:特徴量の説明で「Age:年齢」、「Sex:性別」、「Body mass index:BMI値」、「Average blood pressure:平均血圧」、「S1〜6:血中のある物質の量」です。
https://3pysci.com/python-sklearn-19/
ということでした。
ここで気になるのは、糖尿病の病状に対して、「血中のある物質の量」が僅かながらでも指標になっているのに、その差が僅かすぎて捉え切れていないのではないかということ。
つまりS1〜S6までの数値を二乗してみたり、掛け合わせてみたりして、良い特徴量とならないかということです。
次回はS1〜S6に限らず全ての特徴量を二乗してみて、良い特徴量にできないか、相関マップを使って調べていきたいと思います。
ではでは今回はこんな感じで。

コメント